E tu… di che evotipo sei?
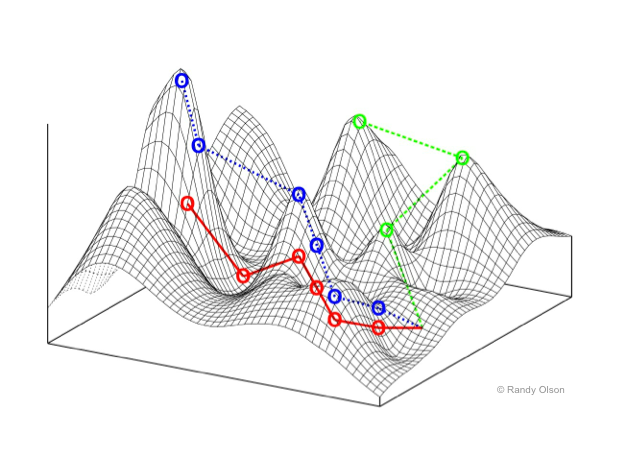
L’evotipo è un concetto utile per capire qual è il più adatto per selezionare in laboratorio una funzione di nostro interesse?
“Facendo ricorso all’evoluzione –ha spiegato Arnold nel discorso per la consegna del Nobel conferitole nel 2018– volevo arrivare a proteine migliori, proteine che rispondessero ai miei scopo (…). L’evoluzione direzionata delle proteine è la ricerca in un nuovo paesaggio adattativo, dove la fitness è definita dai regimi di selezione artificiale da me imposti” (qui il video della conferenza per il Nobel 2018).
Come ben illustrato in un articolo di Francesco Suman su Il Bo Live, l’evoluzione direzionata è un approccio affidabile di ottimizzazione di una funzione: “trova un enzima o una sequenza proteica, ne analizza il rendimento, individua le pressioni selettive da indurre e lascia l’evoluzione lavorare nella direzione stabilita, in cicli di riproduzione e selezione delle varianti desiderate. (…) L’evoluzione direzionata ha prodotto risultati strabilianti nel mondo delle biotecnologie, tra cui nuovi strumenti di editing genetico, nuove terapie enzimatiche, procedure diagnostiche, strumenti per sequenziare e sintetizzare il DNA, metodi di imaging, applicazioni in agricoltura, nel settore del tessile, farmaceutico e industriale, ha persino reso più efficienti i detergenti del nostro bucato”. In questa ottica, la biodiversità dei viventi che ci circonda diventa preziosa anche da un punto di vista pratico in quanto strumento per favorire l’innovazione. Ma come possiamo sfruttare al meglio queste opportunità? Come identificare i modelli sperimentali più utili per favorire l’evoluzione di specifiche funzioni? Per dare una risposta a questo importante quesito, un team di ricercatori dell’Università di Bristol ha sviluppato un approccio (recentemente pubblicato sulla rivista scientifica Nature Communications) che permette di definire a quale evotipo appartengano le specie di potenziale interesse biotecnologico, così da avere uno strumento per capire celermente le potenzialità evolutive di un organismo vivente in laboratorio.
“Capire come ingegnerizzare un vivente in modo efficace con l’evoluzione è una delle sfide più importanti, se non la più grande, che devono affrontare oggi i bioingegneri –ha affermato Claire Grierson, co-autrice della pubblicazione su Nature Communications–. Il nostro lavoro rende disponibile uno strumento di cui vi era una diffusa necessità per descrivere il potenziale evolutivo di un biosistema e di re-immaginare l’ingegneria biologica in modo che sia coerente con le peculiarità evolutive di ciascuna specie”.
Lo strumento ideato dai ricercatori del team inglese sfrutta una soluzione grafica già in uso in biologia, derivata dai paesaggi adattativi (su Pikaia ne abbiamo parlato qui), trasformandola in un paesaggio di potenziale evolutivo. In particolare, per calcolare l’evotipo, il sistema mette a confronto la fitneity (che nasce dalla combinazione fitness e utilità) con la probabilità di evoluzione di uno specifico tratto. Andando a osservare la distribuzione dei picchi (corrispondenti alle funzioni con maggiore potenziale evolutivo in un sistema biologico), possiamo comprendere quali funzioni presentino una evolvibilità, intesa come potenzialità evolutiva, coerente con le funzioni che vogliamo selezionare in laboratorio. Specie simili nel fenotipo, intenso come insieme di proprietà osservabili, possono avere evotipi ben differenti e quindi garantire successi diversi durante esperimenti di evoluzione direzionata. Contrariamente a quanto fatto per decenni nelle biotecnologie, in cui si sono cercate specie chassis, cioè specie che potessero essere usate per le applicazioni più diverse, oggi si vuole capire quale organismo sia meglio per evolvere funzioni di interesse per i ricercatori. L’ingegneria umana e l’evoluzione biologica presentano alcune somiglianze, ma anche significative differenze. Riuscire a definire l’evotipo può quindi essere un modo per usare meglio l’evoluzione come strumento per l’ingegneria.
“Noi alleviamo molecole –ha affermato in una intervista Frances Arnold (qui il testo completo)– come altri allevano cani e gatti. Ho scelto di applicare la selezione artificiale alle proteine, che sono codificate da singoli geni. Con i moderni strumenti di biologia molecolare, possiamo fare tutto in provetta e far evolvere i geni piuttosto che interi organismi. Ciò che è meraviglioso dell’evoluzione è la sua capacità di produrre rapidamente innovazioni: possiamo creare proteine con proprietà sorprendenti e utili in un breve periodo di tempo. Non saremmo in grado di progettare da zero tali proteine, proprio come non siamo in grado di progettare da zero un cavallo da corsa o un barboncino. L’evoluzione ci permette di aggirare la nostra pressoché totale ignoranza su come una sequenza proteica determini i dettagli della sua funzione, dandoci la possibilità di produrre proteine che forse la natura non si è presa la briga di fare”.
Riferimenti: Simeon D. Castle, Claire S. Grierson, Thomas E. Gorochowski (2021) Towards an engineering theory of evolution. Nature Communications 12: e3326.
Fonte immagine: Wikimedia Commons
Biologo e genetista all’Università di Modena e Reggio Emilia, dove studia le basi molecolari dell’evoluzione biologica con particolare riferimento alla citogenetica e alla simbiosi. Insegna genetica generale, molecolare e microbica nei corsi di laurea in biologia e biotecnologie. Ha pubblicato più di centosessanta articoli su riviste nazionali internazionali e tenuto numerose conferenze nelle scuole. Nel 2020 ha pubblicato per Zanichelli il libro Nove miliardi a tavola- Droni, big data e genomica per l’agricoltura 4.0. Coordina il progetto More Books dedicato alla pubblicazione di articoli e libri relativi alla teoria dell’evoluzione tra fine Ottocento e inizio Novecento in Italia.



